对于一个家族或者相似的群体来说,变异往往具有一定的相似性。通过对一个家族或者群体进行联合变异的检测,可以有效提高变异检测的准确率。本文介绍如何通过命令行调用AGS服务API进行群体联合变异检测。
前提条件
开通服务。
背景信息
通过AGS全基因组的变异检测(WGS)生成具备更多未突变点位覆盖信息的GVCF。更多信息,请参见GVCF。通过AGS的merge工具生成多样本的GVCF。更多信息,请参见CombineGVCFs,为后续的变异矫正提供数据支撑。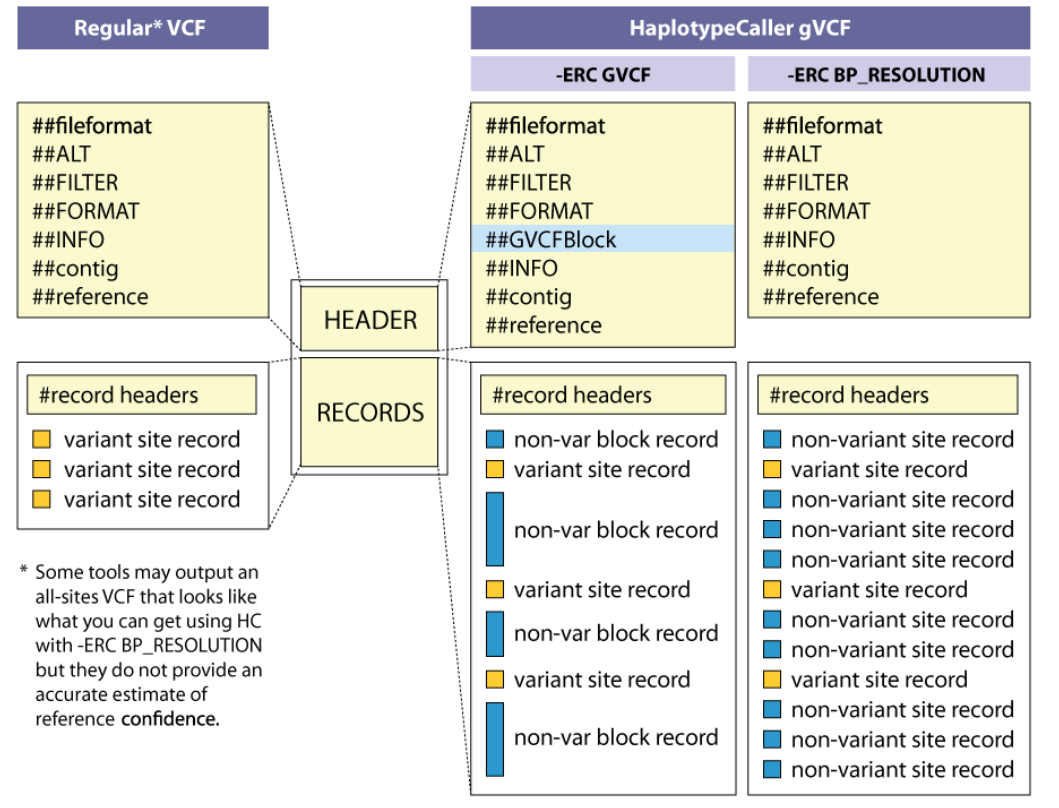

准备工作
- 下载和安装AGS。具体操作,请参见AGS命令行帮助。
- 配置AGS。
ags config init - 确认Bucket的Owner。
- 准备Bucket,并授权AGS服务读写权限和GetBucketInfo权限。说明
- 请确保指定Bucket的Owner是您当前的账号,否则建议您新建一个Bucket后再进行GetBucketInfo的授权。
- 如果您使用的是子账号,需要为子账号授予AliyunOSSFullAccess权限,请参见为RAM用户授权。
- 如果您使用的是子账号,建议您重新创建OSS Bucket,以确保该账号是所使用Bucket的Owner。执行以下命令确认Bucket的Owner。
ossutil stat oss://<your new bucket name>
Usage: ags config oss <your bucket name> e.g. ags config oss my-test-shenzhen - 通过ossutil上传FASTQ或者GVCF数据到OSS Bucket。
关于ossutil的下载和安装,请参见下载和安装ossutil。
说明 目前只支持人类基因数据WGS、WES等,暂不支持甲基化数据,以及动植物数据的比对。Usage: ossutil cp -r <local dir of fastq> <path of oss bucket > e. g. ossutil cp -r ./MGISEQ oss://my-test-shenzhen/MGISEQ
启动群体联合变异检测
- 生成GVCF文件。关于参考基因组--reference,推荐和默认使用
hg19基因组(hs37d5版本)。并且各个样本--reference-group中的SampleName(SM)须为不同,如以下所示,MGISEQ_NA12878_hs37d5_6.gvcf采用的SM为12878,MGISEQ_NA12878_hs37d5_5.gvcf采用的SM为12878_1。说明 如果您已经有生成的GVCF文件,可以跳过该步骤,直接进行群体变异合并检测。Usage: ags remote run wgs \ --region cn-shenzhen # region of oss, e.g. cn-shenzhen, cn-beijing and etc\ --fastq1 MGISEQ/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_1.fq.gz # filename of fastq pair 2, fastq-path\filename \ --fastq2 MGISEQ/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_2.fq.gz # filename of fastq pair 1\ --bucket my-test-shenzhen # Bucket name\ --output-bam bam/MGISEQ_NA12878_hs37d5.bam, # Output BAM to bucket, By default empty, non output of BAM \ --output-gvcf gvcf/MGISEQ_NA12878_hs37d5_5.gvcf # Output filename \ --service "s" #SLA: [n:normal|s:silver|g:gold|p:platinum]\ --reference [hg19|hg38|<reference path on OSS>] # hg19: it is hs37d5 version, GRCh37/hg19 include decoy contig, no support for UCSC hg19. hg38: GRCh38/hg38 include decoy --reference-group "\"@RG\\tID:TEST\\tSM:12878\\tPL:MGISEQ2000\"" # allow to specify reference groups for PL/SM/ID and etc e.g. ags remote run wgs \ --region cn-shenzhen \ --fastq1 MGISEQ/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_1.fq.gz \ --fastq2 MGISEQ/MGISEQ2000_PCR-free_NA12878_1_V100003043_L01_2.fq.gz \ --bucket my-test-shenzhen \ --output-gvcf gvcf/MGISEQ_NA12878_hs37d5_5.gvcf \ --output-bam bam/MGISEQ_NA12878_hs37d5_5.bam \ --service "s" \ --reference hg19 \ --reference-group "\"@RG\\tID:TEST\\tSM:12878_1\\tPL:MGISEQ2000\"" ags remote run wgs \ --region cn-shenzhen \ --fastq1 MGISAMPLE001 \ --fastq2 MGISAMPLE001 \ --bucket my-test-shenzhen \ --output-gvcf gvcf/MGISEQ_NA12878_hs37d5_6.gvcf \ --output-bam bam/MGISEQ_NA12878_hs37d5_6.bam \ --service "s" \ --reference hg19 \ --reference-group "\"@RG\\tID:TEST\\tSM:12878\\tPL:MGISEQ2000\"" - 启动群体变异合并检测。
通过merge调用生产多样本GVCF的合并GVCF cohort.gvcf.gz,相当于GATK CombineGVCFs结果。而联合基因型分析结果output.vcf.gz,相当于GATK GenotypeGVCFs的结果。
Usage: ags remote merge \ --region cn-shenzhen # region of oss, e.g. cn-shenzhen, cn-beijing and etc\ --bucket my-test-shenzhen # Bucket name\ --gvcf gvcf/MGISEQ_NA12878_hs37d5_5.gvcf # filename of gvcf 1\ --gvcf gvcf/MGISEQ_NA12878_hs37d5_6.gvcf # filename of gvcf 2\ --gvcf gvcf/MGISEQ_NA12878_hs37d5_7.gvcf # filename of gvcf 3\ --output-vcf merge/output.vcf.gz # Output filename of vcf, it is simiar to result of GATK GenotypeGVCFs\ --output-gvcf merge/cohort.gvcf.gz # Output filename of gvcf, it is similar to result of GATK CombineGVCFs\ --service "s" #SLA: [s:silver|g:gold|p:platinum]\ e.g. ags remote merge \ --region cn-shenzhen \ --bucket my-test-shenzhen \ --gvcf gvcf/MGISEQ_NA12878_hs37d5_5.gvcf \ --gvcf gvcf/MGISEQ_NA12878_hs37d5_6.gvcf \ --output-vcf merge/output.vcf.gz \ --output-gvcf merge/output.gvcf.gz \ --service "s" INFO[0001] {"JobName":"merge-hck8x"} INFO[0001] Job submit succeed - 列出远程任务。
Usage: ags remote list e.g. ags remtoe list+---------------+-------------------------------+------------+ | JOB NAME | CREATE TIME | JOB STATUS | +---------------+-------------------------------+------------+ | merge-hck8x | 2020-08-31 20:57:06 +0000 UTC | Running | | merge-5c4lt | 2020-08-31 18:01:38 +0000 UTC | Succeeded | +---------------+-------------------------------+------------+
