解密AnalyticDB高效基因检测系统
基于阿里云云原生数据仓库AnalyticDB MySQL版向量分析构建的高效基因检测系统,能够在几十分钟内完成整个病原体的查询检测,大大提升了基因分析性能,为疑难杂症的确诊以及精准治疗提供有力支持。
背景信息
病原体基因检测为医生诊断疑难杂症提供了诊断基础,病原体基因检测流程分为五个步骤,如基因检测流程所示:
采集病人的样本,例如静脉血、痰液、肺泡灌洗液或者脑脊髓液等。
对样本进行培养,然后提取样本中的核酸组织。
通过高通量基因测序仪(High-throughput Sequencing),对核酸序列进行测序。
为保证高通量测序仪的精度,可以将较长的核苷酸序列切成小的分片,分别进行测序。在读取基因序列时,基因序列的数据大小一般为50PB~200PB。
通过高通量测序之后,查询相关病原体标准序列,找到匹配的基因序列。
对切分成片的基因序列进行分析,得到全部基因片段的组成成分即检测结果,从而为疾病的确诊以及精准治疗提供有力支持。
图 1. 基因检测流程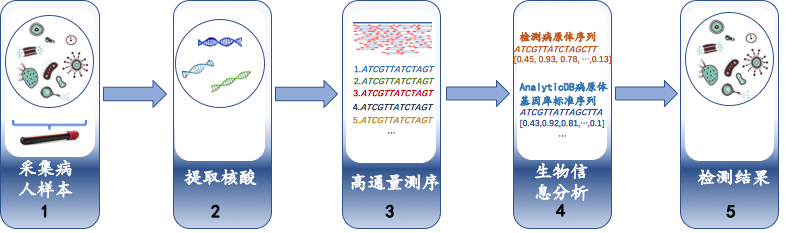
通常在生物分析检测中进行一次病原体检测,大约生成5亿个75BP的基因片段。过滤掉属于人的基因组织序列之后,仍需要查询1亿左右的基因片段。一般可以使用nBlast [1]工具进行基因匹配,整个病原体检测过程大约需要2小时~3小时,耗时较长。
阿里云云原生数据仓库AnalyticDB MySQL版(简称AnalyticDB,原分析型数据库MySQL版)的向量分析提供了一个高效基因检测系统,能够在几十分钟内完成整个病原体的查询检测,大大提升了基因分析性能。
AnalyticDB基因检测功能
系统界面是AnalyticDB基因检测系统的界面。当前系统包含了12182个病毒的碱基序列,病毒的碱基序列切分成150BP的小片段(总共1590804个片段),转化成向量之后存储在AnalyticDB中。
您可以在检测框中输入一段基因序列,通过当前系统进行检测。为方便使用,AnalyticDB基因检测系统自带新型冠状病毒肺炎(简称新冠肺炎)、艾滋病病毒、埃博拉病病毒和中东呼吸综合症基因序列,您可以直接拷贝相关序列,检测系统的查询性能。
图 2. 系统界面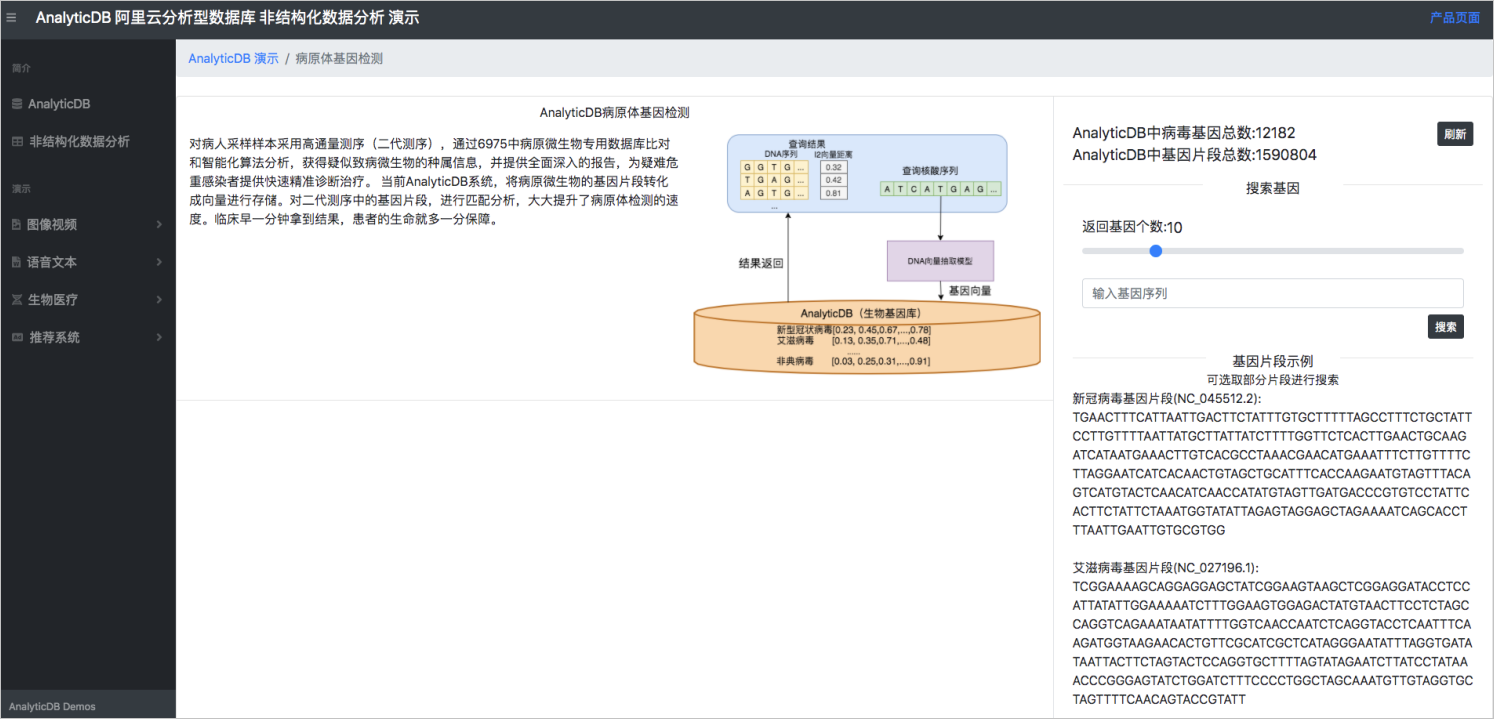
例如某用户在基因序列输入框中输入一段新冠肺炎病毒序列,然后单击检索,系统在毫秒之内返回相关基因片段,查询的基因序列显示某用户要查询的病毒序列,如示例查询所示。
图 3. 示例查询
系统架构
AnalyticDB负责存储和查询基因检测系统中所有结构化数据(例如基因序列的长度、基因的名称、基因的种类以及基因的详细介绍,DNA或者RNA等)和基因序列产生的特征向量。查询时使用基因向量抽取模型,将基因转化成向量,在基因库中进行粗排检测,然后使用经典的Needleman-Wunsch [4]算法在匹配的向量结果集中进行精排,返回最相似的基因序列,如系统架构所示。
图 4. 系统架构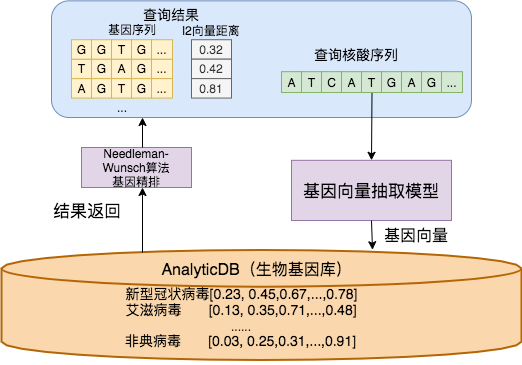
端到端的基因分析
本示例模拟人体的基因采样,将新冠肺炎病毒基因(塞尔维亚MT450872 [2],美国MT450873 [3])和中东呼吸综合症MERS基因(NC_019843.3 [10])三株病毒混合在一起,打散成75BP的序列作为测试集。通过AnalyticDB高效基因检测系统检测分析,能够快速识别当前测试集合中包含新冠肺炎病毒和MERS病毒。
图 5. 基因匹配结果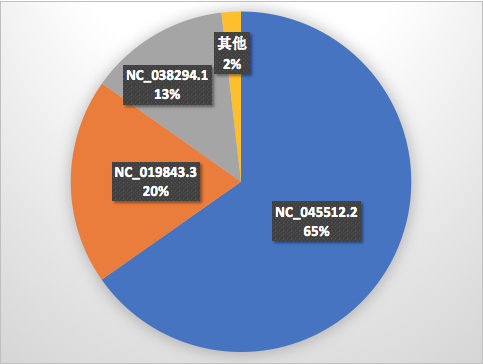
如基因匹配结果所示,AnalyticDB高效基因检测系统从病毒库检测返回三个基因组(NC_045512.2,NC_019843.3和NC_038294.1)。其中NC_045512.2(占比65%)是武汉海鲜市场的新冠肺炎病毒的基因;NC_019843.3(占比20%)是MERS病毒的基因;NC_038294.1(占比13%)是Beta型英国冠状病毒,是MERS病毒的另外一个名字 [8],也属于MERS病毒。通过分析,当前测试样本中包含了新冠肺炎病毒和MERS病毒。
基因查询过程
AnalyticDB基因向量抽取算法通过DNA K-Mer模型得到每个k-mer的向量。如DNA序列转向量所示的一段12BP的基因序列,在这段基因序列中抽取出5个8-mers,然后将这5个8-mers转换成对应的向量,求和归一化之后,就是这段12BP基因序列的向量。为提升精度,也可以使用doc2vec [6]等学习模型对整段基因片段进行转化。详细的基因向量抽取算法请参见基因向量抽取算法。
图 6. DNA序列转向量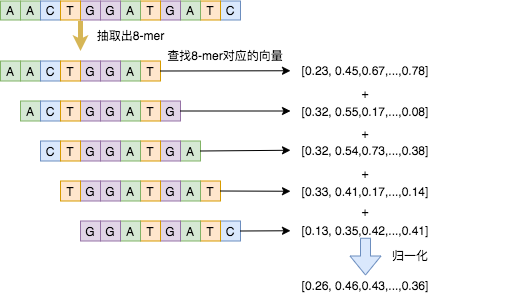
基因精度测试
为测试AnalyticDB高效基因检测系统的基因精度,训练了两个模型,全部病毒模型和21个病原体细菌模型(痤疮丙酸杆菌、金黄色葡萄球菌、表皮葡萄球菌、溶血葡萄球菌、大肠埃希氏菌、鲍曼不动杆菌、结核分枝杆菌、肺炎链球菌、肺炎克雷伯氏菌、流感嗜血杆菌、副流感嗜血杆菌、嗜麦芽窄食单胞菌、铜绿假单胞菌、屎肠球菌、纹带棒状杆菌、人疱疹病毒4型-EB病毒、细环病毒、人腺病毒B组、黄曲霉、白色假丝酵母、耶氏肺孢子菌)。将一个基因,每隔150个BP进行切分,然后将150BP的小分段转换成向量并存入向量库。病毒数据集包括12182个病毒、1590804个分段,21个细菌共275个基因,1521807个分段。
实验一
目的:
随机在当前基因库中取出75BP的小片段,查询75BP的基因片段是从哪个基因的哪个片段中提取的。
结果:
在基因库中检测75BP的基因片段,在返回的前N个结果集中,查看是否包含75BP基因片段对应的基因片段,通过公式计算Top-n的精度(Precision(n)),其中n表示查询返回的列表长度;u表示查询次数,取值为1000次。
图 7. 公式
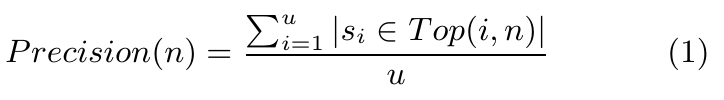
以上公式的含义为在第i次查询中,序列si是否出现在Top-n的列表中,出现为1,不出现为0。如果n越小,精度越高,实际应用中的效率也越高。针对两个模型来说,top20的精度均在99%以上,精度在0.99以上,如实验一结果所示,对于实际检验基因片段是否包含物种基因是足够的。
表 1. 实验一结果
数据集
top1
top2
top3
top4
top5
top10
top20
病毒
0.866
0.965
0.983
0.986
0.99
0.992
0.994
21个细菌
0.901
0.975
0.987
0.987
0.993
0.994
1.0
实验二
目的:
随机在当前基因库中取出75BP的基因片段,将75BP的基因段进行2%的随机突变,在基因库中查询是否包含75BP的基因片段对应的基因片段。
说明自然界中基因突变的概率很低,例如人的30亿个碱基,新生儿会有30个基因发生突变。病毒RNA的突变概率会高一些,一般也小于1%
结果:
在基因库中检测随机突变后的75BP基因段,在返回的前N个结果集中,找到了包含75BP的基因片段对应的基因片段。基因突变之后,虽然查询精度有所下降,但top20的精度也都达到了0.99,如实验二结果所示。
表 2. 实验二结果
数据集
top1
top2
top3
top4
top5
top10
top20
病毒
0.846
0.954
0.960
0.976
0.98
0.982
0.99
21个细菌
0.884
0.961
0.968
0.973
0.973
0.989
1.0
实验三
目的:
对比在AnalyticDB数据库和Blast数据库中的基因检测速度。
结果:
为得到实验结果下载了病毒序列、菌类基因序列、部分植物基因序列 [7],总数量为9.7GB。分别将相关数据导入AnalyticDB数据库和Blast数据库中,分别进行100次不同的查询,取实验结果的平均值。Blast需要3.22秒才能返回结果,而AnalyticDB在算法精度为top30、精度为0.95的准确性下,测试端到端的查询(包括查询基因转换向量、向量粗排和Needleman-Wunsch算法的精排)只需要0.257秒,相比Blast的3.22秒,AnalyticDB提升了12.5倍,如实验三结果所示。
表 3. 实验三结果
算法
响应时间(秒)
Blast
3.27
AnalyticDB
0.257
附录
[1] blast+ https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/.
[4] Needleman, Saul B. & Wunsch, Christian D. (1970). "A general method applicable to the search for similarities in the amino acid sequence of two proteins". Journal of Molecular Biology. 48 (3): 443–53. doi:10.1016/0022-2836(70)90057-4. PMID 5420325.
[5] Mikolov Tomas; et al. (2013). "Efficient Estimation of Word Representations in Vector Space". arXiv:1301.3781.
[6] 基因数据集 https://www.ncbi.nlm.nih.gov/genome/viruses/variation/help/flu-help-center/ftp/.
[7] de Groot RJ Baker SC Baric RS et al. Middle East respiratory syndrome coronavirus (MERS-CoV): announcement of the Coronavirus Study Group. J Virol. 2013; 87: 7790-7792.
[8] NC_045512.2.
[9] NC_019843.3.
[10] NC_038294.1.
