基于E-HPC Next Alphafold3集群模板,一键创建集群并部署Afusion Web服务,并基于Afusion实现Alphafold3蛋白质复合物结构预测作业的提交及结果查看实践说明。
准备操作
Alphafold3集群模板目前仅支持上海和北京地域。
E-HPC架构组网
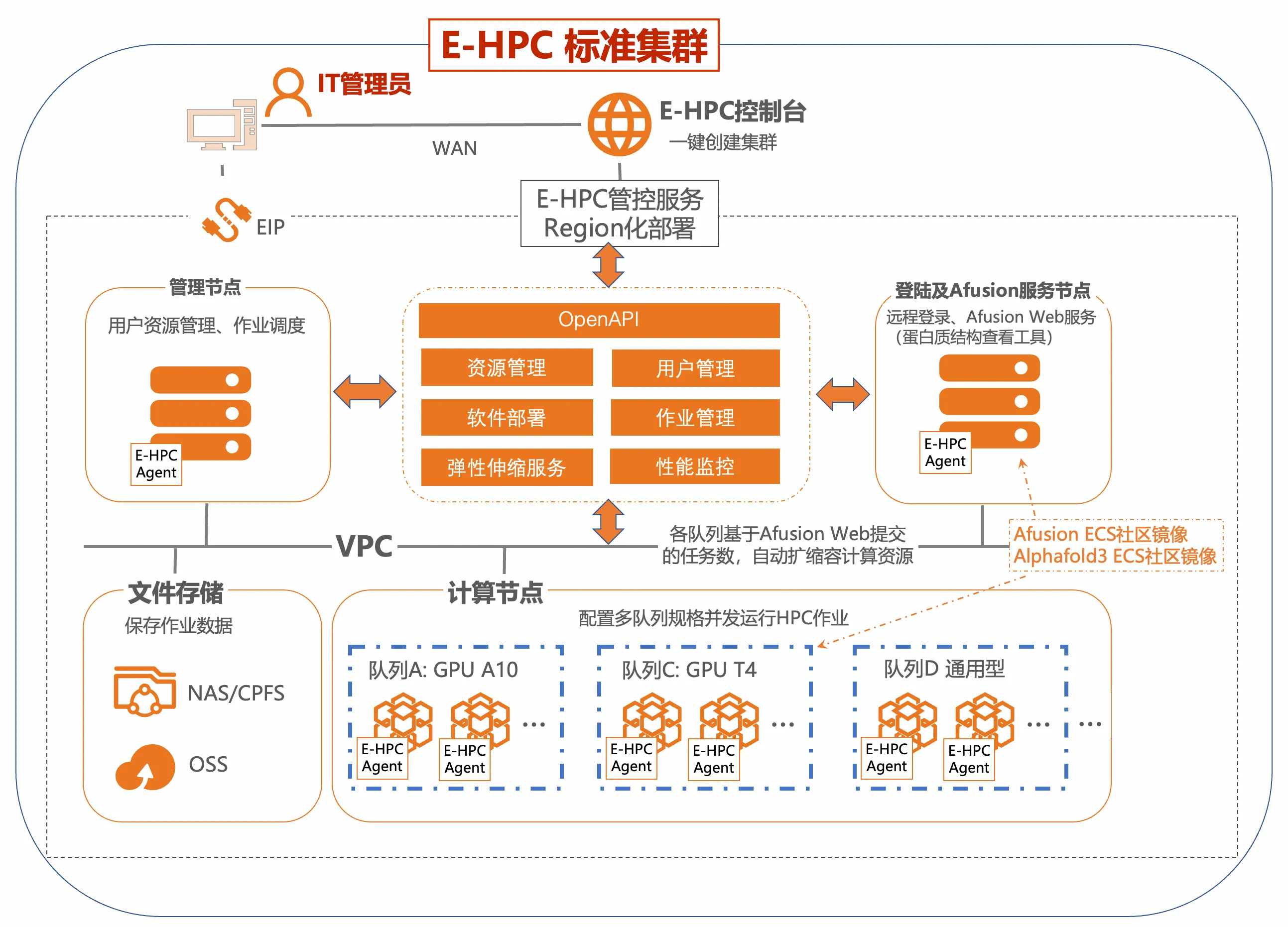
图示集群的网络架构。此架构基于E-HPC Next Alphafold3模板创建。它主要包含三部分:管理节点、计算节点队列和登录节点。登录节点承载Afusion服务。所有节点均自动挂载NAS文件存储。节点与存储资源位于同一VPC网络。若需本地连接,可按需配置公网EIP。
创建集群
进入集群列表页面。
登录弹性高性能计算控制台。
在顶部菜单栏左上角处,选择地域。
在左侧导航栏,单击集群。
在集群列表页面,单击集群模板。在弹出的对话框中,选择Alphafold3。

确认集群配置,然后单击创建集群,完成集群的创建。
说明如果创建集群时提示规格无库存,请暂时关闭队列自动伸缩。这能绕开临时资源短缺,确保集群成功部署。
为增强系统安全性并隔离不同用户的数据,用户管理添加一个具备
sudo权限的新用户,以避免直接使用root账户进行操作。(可选)您可以通过配置集群自动伸缩节点,实现计算节点的动态分配,无需手动操作。
AF3算例执行
数据准备
示例使用的模型参数,您可以通过官网申请。
Afusion提交Alphafold3任务
Afusion页面登录。
进入集群列表页面。
登录弹性高性能计算控制台。
在顶部菜单栏左上角处,选择地域。
在左侧导航栏,单击集群。
在集群页面,找到目标集群,单击右上角的远程连接。在远程连接对话框中,您可以查看集群登录节点的公网IP地址。

登录Afusion页面
http://<集群登录节点的公网IP>:8501。说明页面无法访问,通常是安全组未放行
8501端口。进入集群详情页,切换到集群配置,单击安全组ID放行该端口。
设置与作业相关的信息(Job Settings)。
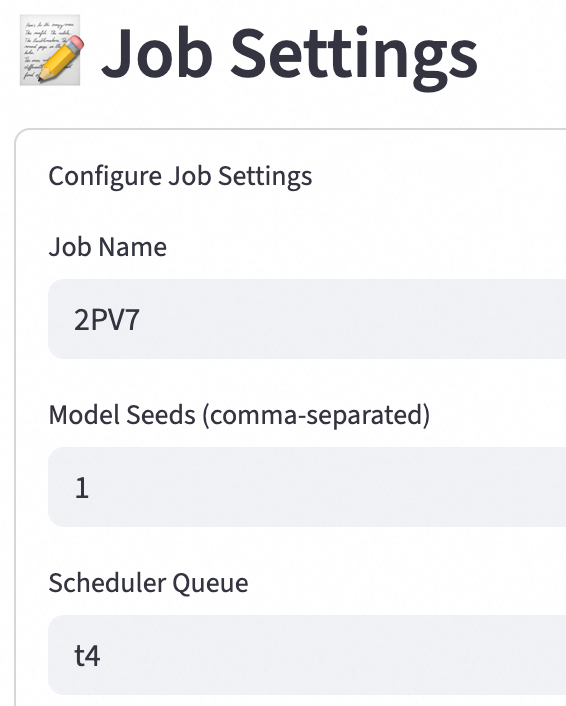
关键参数:
Model Seeds (comma-separated):可以在这里输入一个或多个数字,用逗号分隔,这些数字将作为模型的随机种子,用于确保模型预测的可重复性。不同的种子可能会导致模型产生不同的预测结果。
Scheduler Queue:调度队列,可以在这里指定作业提交到的调度队列(例如:t4)。
在此输入待预测的生物序列 (Sequence)。若无自有序列,可加载开源算例进行体验。
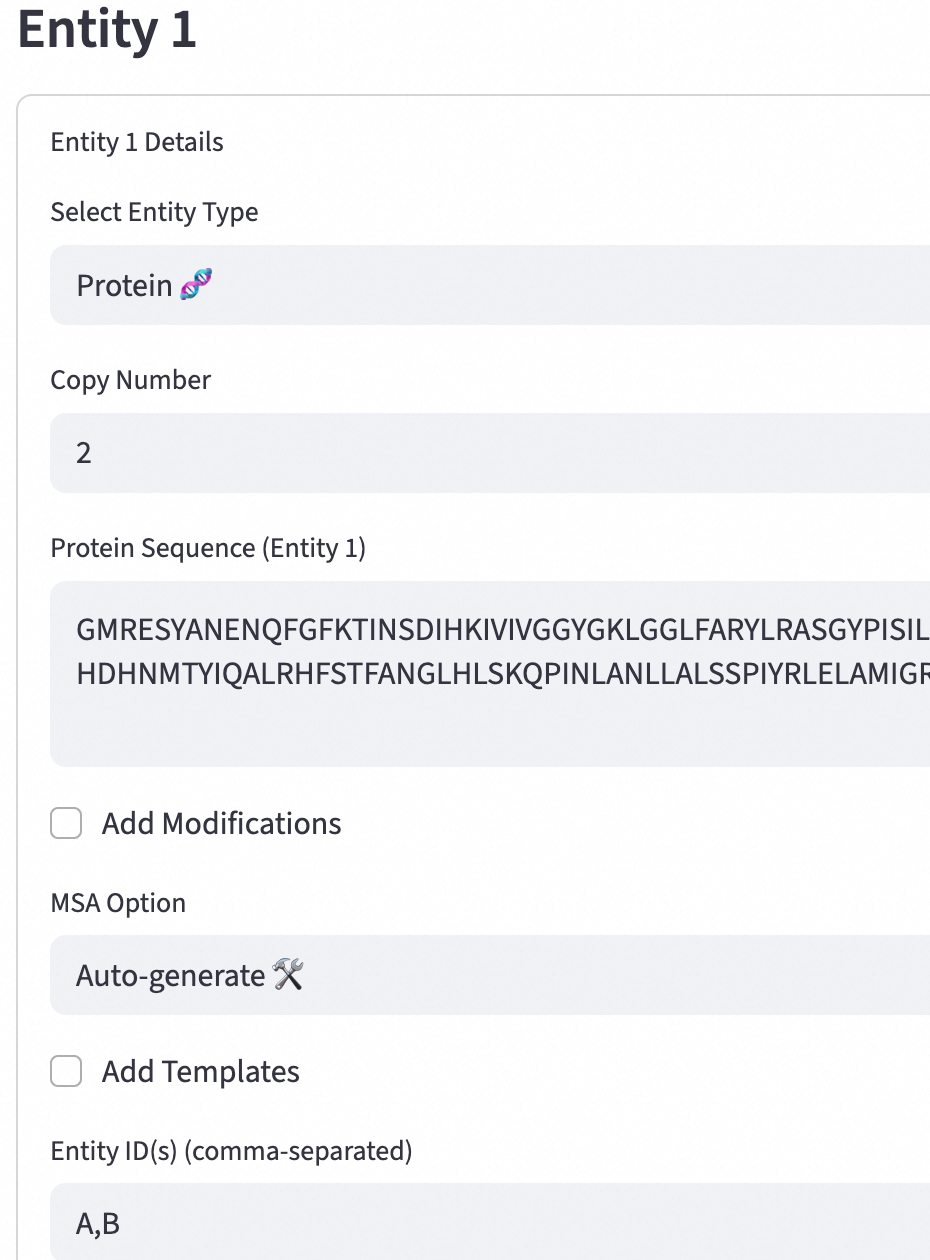
关键参数:
Protein Sequence (Entity 1):输入实体的蛋白质序列,这是进行结构预测的基础数据。
Entity ID(s) (comma-separated):每个实体唯一的标识符,用于在数据库中查找和引用。
配置运行AlphaFold 3预测所需的参数并运行。

关键参数:
AF Input Path:指定存放输入数据(如蛋白质序列、MSA等)的目录。
如:/home/test/af_input
AF Output Path:指定预测结果将保存在这个目录。
如:/home/test/af_output
Model Parameters Directory:指定模型参数文件的路径。
将数据准备阶段申请的模型参数,存放于集群共享目录,如:/opt/data 或/home/data。
Databases Directory:指定数据库文件的路径。AlphaFold 3在进行预测时需要使用一些数据库(如UniRef、Pfam等)来进行多序列比对(MSA)等操作。
E-HPC Alphafold3集群模板中已包含,默认路径:
/data/af3_databases
设置完成后,单击Run AlphaFold 3 Now执行任务。
查看执行日志。

查看任务执行详情。
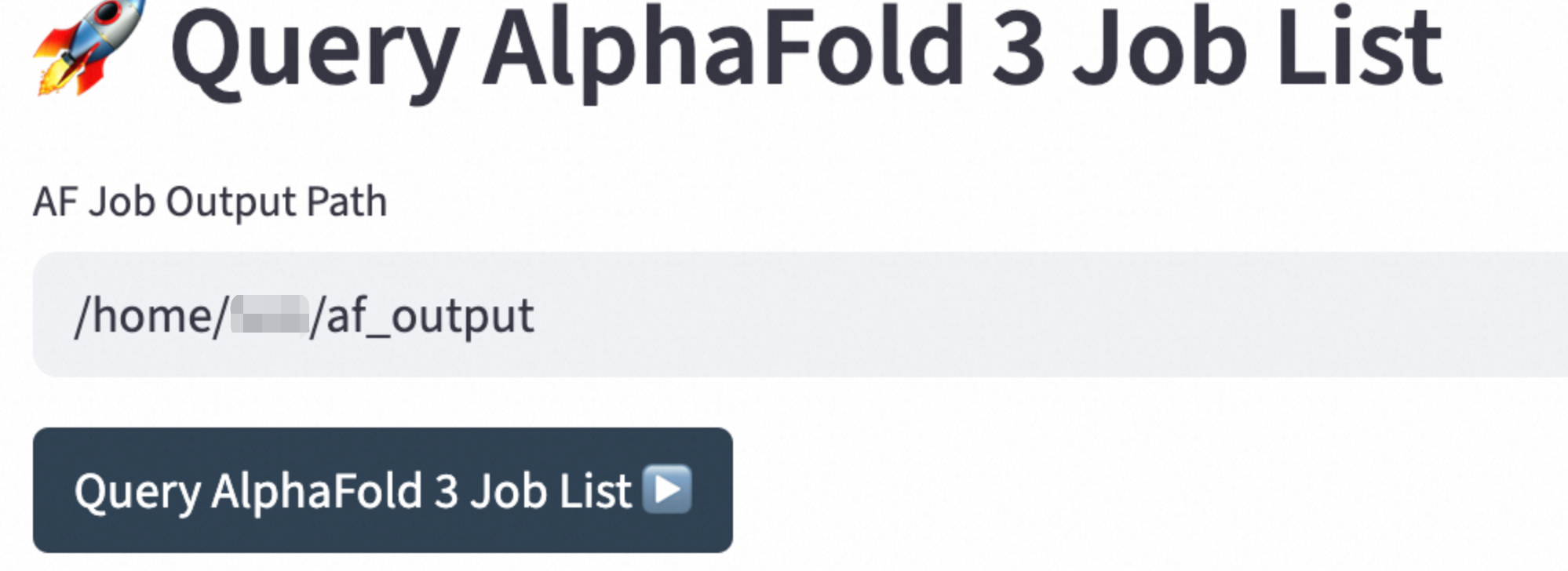
在Query AlphaFold 3 Job List,输入预测结果输出目录,单击Query AlphaFold 3 Job List,查询文件列表。
在Query AlphaFold 3 Job Detail,输入查询到的文件名,单击Query AlphaFold 3 Job Detail查询。
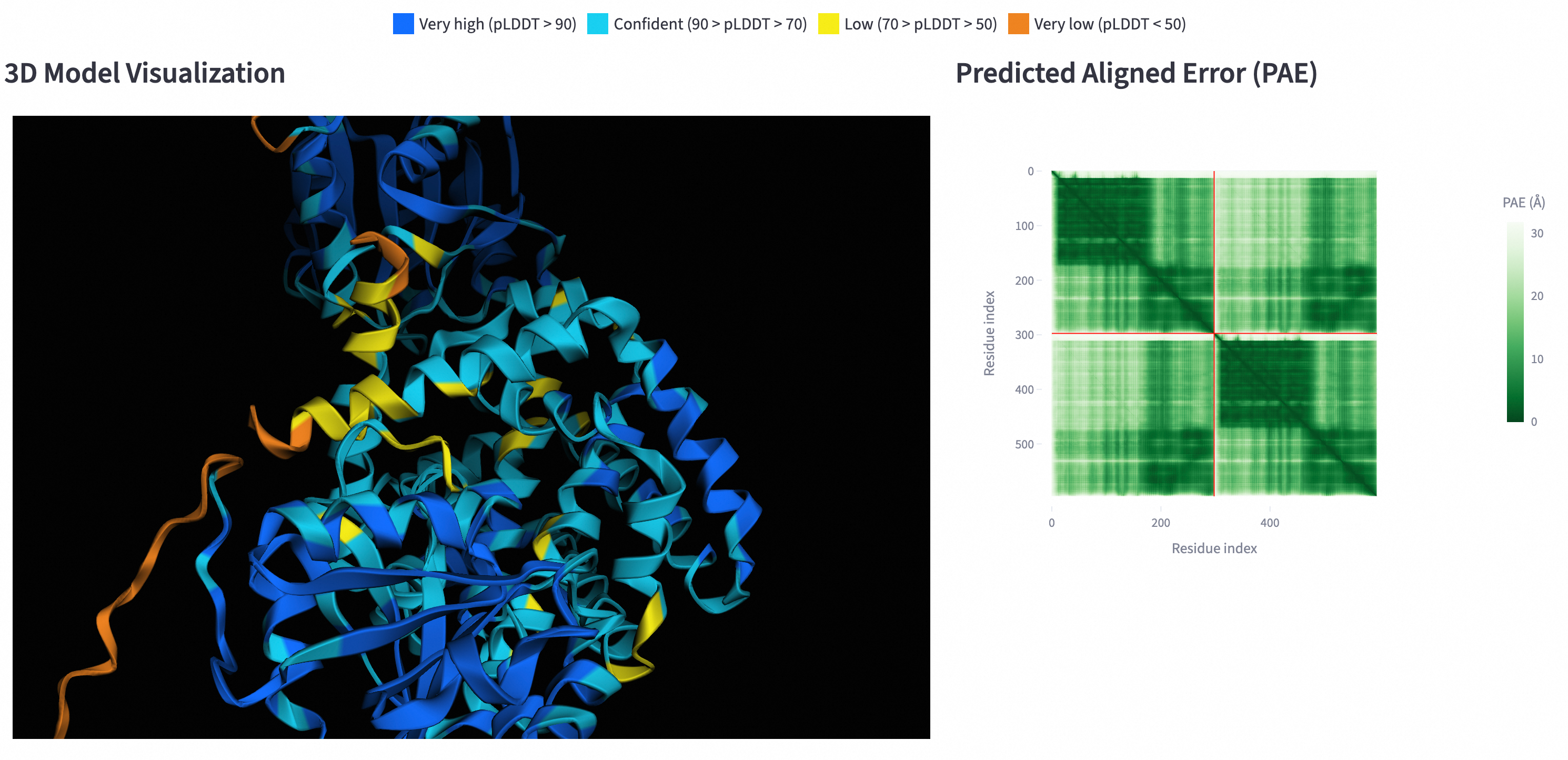
查看可视化结果。
说明图形无法显示,可能是浏览器禁用了图形加速。在Chrome中,进入,然后开启图形加速。
结果数据下载。
my_alphafold_job_model.cif是Alphafold3输出的主要蛋白质三维结构文件。请在Afusion Web 的3D Model Visualization页面查看,或使用VMD、PyMOL等本地软件打开分析。
性能参考
测试场景:2PV7 嗜热菌合成酶蛋白质折叠预测。
根据该场景的测试数据,推荐以下规格:
成本优先:推荐ecs.gn7i-c8g1.2xlarge(8c30g)。
性能优先:推荐ecs.gn7i-2x.8xlarge。
此数据仅供参考,不构成性能承诺。实际性能可能因具体环境、硬件和网络状况而异。
规格 | GPU | Reference数据存储 | inference时长(min) | 样本数/小时 |
ecs.gn7i-c8g1.2xlarge(8c30g) | A10 | ESSD PL0 | 31.7 | 1.89 |
ecs.gn7i-c8g1.2xlarge(8c30g) | A10 | NAS | 40.5 | 1.48 |
ecs.gn6v-c8g1.2xlarge(8c32g) | V100 | ESSD PL0 | 35 | 1.71 |
ecs.gn6i-c8g1.2xlarge(8c31g) | T4 | ESSD PL0 | 51.8 | 1.16 |
ecs.gn7i-2x.8xlarge(32c128g) | A10 | ESSD PL3 | 7.7 | 7.81 |
