高效基因序列检索助力快速分析肺炎病毒
云原生数据仓库 AnalyticDB MySQL 版是云端托管的PB级高并发低延时数据仓库,通过AnalyticDB for MySQL向量检索功能构建的基因检索系统,支持毫秒级针对10亿级别的向量数据进行查询分析,更加快速、高效的为肺炎病毒防控、研发治疗药物以及相关疫苗提供帮助。
基因序列检索技术应用范围和现状
基因序列检索技术主要应用于以下场景:
用于肺炎病毒的溯源和分析,找到病毒宿主,做好有效防范。
用于分析肺炎病毒的复制和传播过程,为研发治疗药物和疫苗提供帮助。
用于检索与肺炎病毒相似的病毒基因序列。
当前的基因匹配算法太慢,迫切需要高效匹配算法进行基因序列检索。阿里云AnalyticDB for MySQL团队将基因序列片段转化成对应的1024维特征向量,将两个基因片段的匹配问题,转换成了两个向量间的距离计算问题,从而大大降低了计算开销,实现毫秒级返回相关基因片段,完成基因片段的首次筛选。然后,使用基因相似计算BLAST算法,完成基因相似度的精确排查,从而高效率完成基因序列的匹配计算。匹配算法从原来O(M+N)的复杂度降低到O(1)。同时,阿里云AnalyticDB for MySQL提供强大的机器学习分析工具,通过基因转向量技术,将局部的和疾病相关的关键靶点基因片段转成特征向量,用于基因药物的研发,大大加速了基因分析过程。
AnalyticDB for MySQL基因检索系统
肺炎病毒的RNA序列可以用一串核酸序列(又称碱基序列)表示,RNA序列含有四种核苷酸,分别用A、C、G和T表示,分别代表腺嘌呤、胞嘧啶、鸟嘌呤、胸腺嘧啶。每个字母代表一种碱基,无间隔排列在一起。每一个物种的RNA序列均不相同但又有规律,基因检索系统可以通过输入一串病毒的基因片段,检索相似的基因,用来对病毒的RNA序列进行分析。
为方便演示AnalyticDB for MySQL基因片段检索方法,我们从GenBank下载了大量病毒的RNA片段,并将GenBank内部关于病毒的论文以及Google Scholar中相关病毒的论文导入AnalyticDB for MySQL基因检索数据库中。
AnalyticDB for MySQL会将肺炎病毒的序列上传到AnalyticDB for MySQL基因检索系统中,AnalyticDB for MySQL基因检索系统只需几毫秒即可检索到相似的基因片段(当前示例系统只返回匹配度超过0.8的基因片段)。从返回的基因片段得出穿山甲携带的肺炎病毒(GD/P1L)、蝙蝠携带的肺炎病毒(RaTG13)以及SARS和MARS病毒,其中GD/P1L的序列匹配度最高为0.974,由此推断出肺炎病毒很可能是通过穿山甲传染到人的。
如果RNA片段非常相似,说明这两个RNA可能有相似的蛋白质表达和结构。通过基因检索工具,可以看到SARS和MARS与肺炎病毒的匹配度为0.8以上,说明可以将一些SARS或者MARS的研究成果应用到肺炎病毒上。系统提取了每种病毒的论文,通过文本分类算法,将论文划分为检测类、疫苗类和药物类。其中,对SARS有效的荧光定量PCR检测,目前正应用于肺炎病毒的检测;基因疫苗的方法以及诱导体内免疫疫苗的方法,也正在展开研究;治疗药物中瑞德西韦以及相关的干扰素也都用于肺炎病毒的治疗上。
实现架构
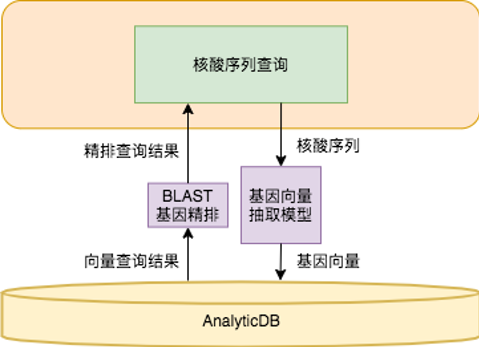
AnalyticDB for MySQL基因检索系统中,AnalyticDB for MySQL负责存储和查询所有结构化数据(例如基因序列的长度,长度包含基因的论文名称、基因种类、DNA或者RNA等)和基因序列产生的特征向量。查询数据时,通过基因向量抽取模型将基因转化成向量,在AnalyticDB for MySQL向量数据库中进行粗排检索,然后在返回的向量匹配结果集中使用经典的BLAST算法进行精确检索,返回最相似的基因序列。
AnalyticDB for MySQL基因检索系统的核心是基因向量抽取模型,该模块可以将核苷酸序列转化成向量。目前AnalyticDB for MySQL抽取了各种病毒的RNA全部序列样本进行训练,可以非常方便的对病毒的RNA进行相似度计算。同时,基因向量抽取模型也可以扩展应用于其他物种基因检索。
基因向量抽取算法
目前词向量技术已经非常成熟,被广泛应用于机器翻译、阅读理解、语义分析等相关领域,并取得了巨大成功。词向量化采用了分布式语义的方法表示一个词的含义,一个词的含义就是这个词所处的上下文语境。例如高中英语中的完形填空题,一篇短文空出10个地方,学生根据空缺词的上下文语境选择合适的词。也就是说上下文语境能够准确的表达这个词,如果某位同学选择了正确的选词,表示该同学理解了空缺词的含义。因此,通过上下文词的关系,采用词向量算法,可以为每个词生成一个向量,通过计算两个词向量之间的相似度,得到两个词的相似度。
同样的道理,基因序列的排列具有一定的规律,并且每一部分基因序列所表达的功能和含义不同。可以将很长的基因序列划分成小的单元片段(也就是词)进行分析,并且这些词也有上下文语境,这些词相互连接、相互作用共同完成相对应的功能,形成合理的表达。因此,生物科学家们采用词向量算法对基因序列单元进行向量化,两个基因单元相似度很高,说明需要这两个基因单元共同来表达和完成相应的功能。
总体而言,AnalyticDB for MySQL基因向量抽取算法分为三步:
在氨基酸序列中定义词
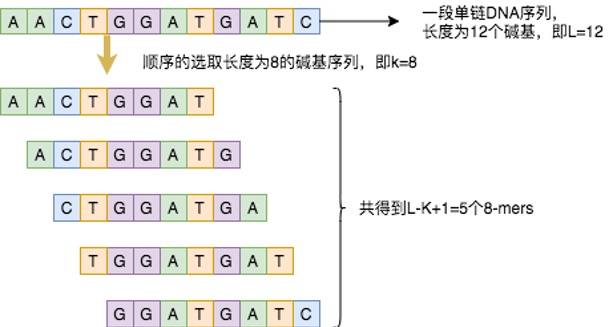
生物信息学中用K-mers来分析氨基酸序列,k-mer是指将核酸序列分成包含k个碱基的字符串,即从一段连续的核酸序列中迭代选取长度为K个碱基的序列,若核酸序列长度为L,k-mer长度为K,那么可以得到L-K+1个k-mers。例如下图所示,假设某序列长度为12,设定选取的k-mer长度为8,则得到(12-8+1=5)5个5-mers。这些k-mer,就是氨基酸序列中词。
找到氨基酸序列的上下文语境,将基因序列中的词转换成1024维向量。
对于词向量算法而言,另一个重要的问题就是上下文的语境。AnalyticDB for MySQL基因向量抽取算法在氨基酸片段中选择一个长度为L的窗口,该窗口内的氨基酸片段可认定为在同一语境内。例如CTGGATGA是一段核酸序列,选取了长度为10的窗口,AnalyticDB for MySQL基因向量抽取算法将CTGGATGA转换成5个5-mers即{AACTG, ACTGG, CTGGA, GGATG, GATGA}。对于其中一个5-mer {CTGGA}而言,另外四个{AACTG, ACTGG, GGATG, GATGA}5-mers就是当前5-mer {CTGGA} 的上下文语境。AnalyticDB for MySQL基因向量抽取算法套用词向量空间训练模型,对已有生物基因的k-mers进行训练,便可将一个k-mer(基因序列中的一个词)转换成1024维向量。
类似于词向量模型,k-mer向量模型也拥有和词向量模型相似的数理计算性质。
向量减法:
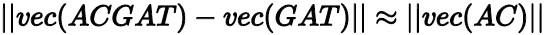
向量加法:

向量减法公式说明核苷酸序列ACGAT的向量减去GAT序列的向量和AC的序列向量距离非常接近。向量加法公式说明核苷酸序列AC的向量加上ATC序列的向量和ACATC序列的向量距离也很接近。因此,根据这些数理特征,计算一个长氨基酸序列向量时,可以将这个序列中每一段的k-mer序列进行累加,最后进行归一化就能得到整个氨基酸序列的向量。同时,为提升精度,可以将基因片段看作一个文本,使用doc2vec函数将整个序列转换成向量进行计算。为进一步验证算法性能,AnalyticDB for MySQL基因向量抽取算法计算了常用于基因检索库中的BLAST[6]算法序列与基因转向量l2距离序列的相似度,两个序列的斯皮尔曼等级相关系数是0.839。 以上得出结论,将DNA序列转换成向量用于相似基因片段的初次筛选,是有效且可行的。
向量检索功能概述
一般包含向量检索的应用系统中,开发者通常会使用向量检索引擎(例如Faiss)存储向量数据,然后使用关系型数据库存储结构化数据。因此,查询时也需要交替查询两个系统,明显额外增加了开发人员的工作量,数据查询性能也不是最优。
AnalyticDB for MySQL是云端托管的PB级高并发低延时数据仓库,可以毫秒级针对10亿级别的向量数据进行查询,100毫秒级别的响应时间。AnalyticDB for MySQL全面兼容MySQL协议以及SQL:2003 语法标准,其向量检索功能支持对图像、文本推荐、声纹、核苷酸序列等相似性进行查询和分析,目前在多个城市的安防项目中已大规模部署了AnalyticDB for MySQL。
AnalyticDB for MySQL支持结构化和非结构化数据的近似检索和分析,通过SQL接口即可快速搭建基因检索或者基因+结构化数据混合检索等系统。在混合检索场景中AnalyticDB for MySQL的优化器会根据数据的分布和查询条件选择最优执行计划,在保证数据召回率的同时,得到最优的性能。例如,通过以下一条SQL即可检索RNA核酸序列。
-- 查找RNA和提交的序列向量相近的基因序列。
select title, # 文章名
length, # 基因长度
type, # mRNA或DNA等
l2_distance(feature, array[-0.017,-0.032,...]::real[]) as distance # 向量距离
from demo.paper a, demo.dna_feature b
where a.id = b.id
order by distance; # 用向量相似度排序上述SQL中表demo.paper用于存储上传的每篇文章的基本信息,demo.dna_feature存储各个物种的基因序列对应的向量。通过基因转向量模型,将要检索的基因转成向量[-0.017,-0.032,...],然后在AnalyticDB for MySQL数据库中进行检索。
当前系统也支持结构化信息+非结构化信息(核苷酸序列)的混合检索,例如查找和冠状病毒相关的类似基因片段时,只需要在SQL中增加where title like'%COVID-19%' 即可。
附录
[1] Mikolov Tomas; et al. (2013). "Efficient Estimation of Word Representations in Vector Space". arXiv:1301.3781.
[2] Mikolov Tomas, Sutskever Ilya, Chen Kai, Corrado, Greg S. and Dean Jeff (2013). Distributed representations of words and phrases and their compositionality. Advances in Neural Information Processing Systems. arXiv:1310.4546. Bibcode:2013arXiv1310.4546M.
[3] Mapleson Daniel, Garcia Accinelli, Gonzalo, Kettleborough George, Wright Jonathan and Clavijo, Bernardo J. (2016). "KAT: a K-mer analysis toolkit to quality control NGS datasets and genome assemblies". Bioinformatics. 33(4): 574–576. doi:10.1093/bioinformatics/btw663. ISSN 1367-4803. PMC 5408915. PMID 27797770.
[4] Quoc Le and Tomas Mikolov. (2014). Distributed representations of sentences and documents. In International Conference on Machine Learning, pages 1188–1196.
[5] 人类基因组hg38, https://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.chromFa.tar.gz.
[7] Julia Piantadosi, Phil Howlett and John Boland. (2007). "Matching the grade correlation coefficient using a copula with maximum disorder", Journal of Industrial and Management Optimization, 3 (2), 305–312.
[8] Stephen Woloszynek, Zhengqiao Zhao, Jian Chen and Gail L. Rosen. (2019). "16s rRNA sequence embeddings: Meaningful numeric feature representations of nucleotide sequences that are convenient for downstream analyses", PLoS Computational Biology, 15(2), e1006721.
[9] James K. Senter, Taylor M. Royalty, Andrew D. Steen and Amir Sadovnik. (2019) "Unaligned Sequence Similarity Search Using Deep Learning.", arXiv e-prints.
[10] Ng Patrick. (2017) dna2vec: consistent vector representations of variable-length k-mers. arXiv preprint, arXiv:1701.06279.