本文介绍基于Tair Vector向量引擎实现化合物或药物分子结构近似检索的解决方案。
背景信息
向量(Vector)检索在AI制药中扮演着至关重要的角色,在该方案中,通常以向量表示化合物和药物,并通过向量空间中的相似度计算来预测、优化它们之间的相互作用。这种方案可以快速地筛选出具有优秀相互作用的化合物和药物,从而加速新药的研发进程。同时,向量检索也能够提高药物筛选的准确度和效率,为医药研究人员提供更加高效、精确的药物研究方法。
相比较传统的向量检索服务,Tair Vector的所有数据均在内存中,支持实时更新索引,具有更短的读写时延。同时,Vector提供的近邻查询(TVS.KNNSEARCH等)支持从数据库中高效地获取与目标分子结构最为相似的Top k个分子结构(k支持自定义),可降低人工误操作或漏操作对项目造成不可控的风险。
方案概述
整体流程图如下。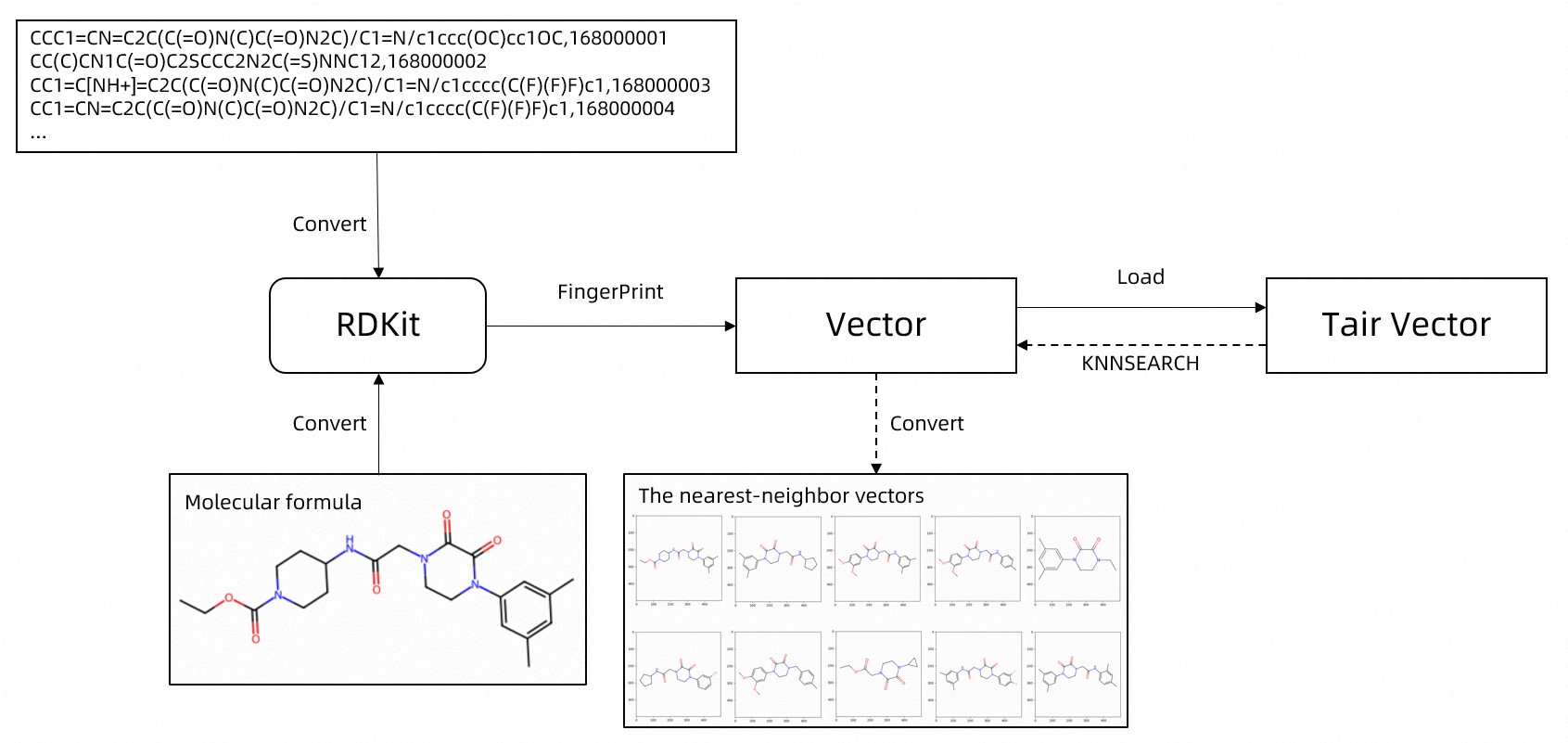
下载化学分子数据集。
本示例使用的测试数据为PubChem开源数据集,共计11012行数据,上述下载地址提供的数据为SMI格式,数据示例如下,两列分别为化学分子式和唯一ID。
说明在实际项目中,您可以写入更多数据,体验Tair毫秒级别检索的特性。
CCC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1ccc(OC)cc1OC,168000001 CC(C)CN1C(=O)C2SCCC2N2C(=S)NNC12,168000002 CC1=C[NH+]=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1cccc(C(F)(F)F)c1,168000003 CC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1cccc(C(F)(F)F)c1,168000004若您从官方下载,则数据为SDF格式,您还需通过下述代码将其转换为SMI格式:
连接Tair实例,具体实现可参见示例代码中的
get_tair函数。在Tair中创建用于存储分子结构的向量索引,具体实现可参见示例代码中的
create_index函数。写入分子结构示例数据,具体实现可参见示例代码中的
do_load函数。将通过RDKit库提取分子结构数据的向量特征,并通过Vector的TVS.HSET命令,将其唯一ID、特征信息和化学分子式存入Tair中。
进行相似分子结构查询,具体实现可参见示例代码中的
do_search函数。将通过RDKit库提取待查询分子结构的向量特征,然后通过Vector的TVS.KNNSEARCH命令,在Tair的指定索引中,查询与该目标最为相似的分子结构。
示例代码
本示例的Python版本为3.8,需提前安装如下依赖库:pip install numpy rdkit tair matplotlib。
import os
import sys
from tair import Tair
from tair.tairvector import DistanceMetric
from rdkit.Chem import Draw, AllChem
from rdkit import DataStructs, Chem
from rdkit import RDLogger
from concurrent.futures import ThreadPoolExecutor
RDLogger.DisableLog('rdApp.*')
def get_tair() -> Tair:
"""
连接Tair实例。
* host:Tair实例连接地址。
* port:Tair实例的端口号,默认为6379。
* password:Tair实例的密码(默认账号)。若通过自定义账号连接,则密码格式为“username:password”。
"""
tair: Tair = Tair(
host="r-bp1mlxv3xzv6kf****pd.redis.rds.aliyuncs.com",
port=6379,
db=0,
password="Da******3",
)
return tair
def create_index():
"""
创建用于存储分子结构的向量索引:
* 本示例的索引名称为"MOLSEARCH_TEST"。
* 向量维度为512。
* 计算向量距离函数为L2。
* 索引算法为HNSW。
"""
ret = tair.tvs_get_index(INDEX_NAME)
if ret is None:
tair.tvs_create_index(INDEX_NAME, 512, distance_type=DistanceMetric.L2, index_type="HNSW")
print("create index done")
def do_load(file_path):
"""
您需要输入分子结构数据集的路径,该方法会自动提取分子结构的向量特征(smiles_to_vector),并将数据写入Tair Vector中。
同时,该方法会调用parallel_submit_lines、handle_line、smiles_to_vector、insert_data等函数。
存入Tair的格式为:
* 向量索引名称为“MOLSEARCH_TEST”。
* Key为分子结构的唯一ID,例如“168000001”。
* 特征信息为512维向量信息。
* “smiles”为化学分子式,例如“CCC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1ccc(OC)cc1OC”。
"""
num = 0
lines = []
with open(file_path, 'r') as f:
for line in f:
if line.find("smiles") >= 0:
continue
lines.append(line)
if len(lines) >= 10:
parallel_submit_lines(lines)
num += len(lines)
lines.clear()
if num % 10000 == 0:
print("load num", num)
if len(lines) > 0:
parallel_submit_lines(lines)
print("load done")
def parallel_submit_lines(lines):
"""
并发写入的调度方法。
"""
with ThreadPoolExecutor(len(lines)) as t:
for line in lines:
t.submit(handle_line, line=line)
def handle_line(line):
"""
单个分子结构的写入处理。
"""
if line.find("smiles") >= 0:
return
parts = line.strip().split(',')
try:
ids = parts[1]
smiles = parts[0]
vec = smiles_to_vector(smiles)
insert_data(ids, smiles, vec)
except Exception as result:
print(result)
def smiles_to_vector(smiles):
"""
提取分子结构的向量特征,从SMI格式转换为Vector。
"""
mols = Chem.MolFromSmiles(smiles)
fp = AllChem.GetMorganFingerprintAsBitVect(mols, 2, 512 * 8)
hex_fp = DataStructs.BitVectToFPSText(fp)
vec = list(bytearray.fromhex(hex_fp))
return vec
def insert_data(id, smiles, vector):
"""
将分子结构的Vector写入Tair Vector中。
"""
attr = {'smiles': smiles}
tair.tvs_hset(INDEX_NAME, id, vector, **attr)
def do_search(search_smiles,k):
"""
您需要输入待查询分子结构,该方法会在Tair的指定索引中,查询并返回与该目标最为相似的k个分子结构。
先提取待查询分子结构的向量特征,再通过TVS.KNNSEARCH命令查询到k个(本示例为10)最近的分子结构唯一ID,然后通过TVS.HMGET命令查询到对应的分子式。
"""
vector = smiles_to_vector(search_smiles)
result = tair.tvs_knnsearch(INDEX_NAME, k, vector)
print("与查询目标分子结构最为相似的10个分子结构如下:")
for key, value in result:
similar_smiles = tair.tvs_hmget(INDEX_NAME, key, "smiles")
print(key, value, similar_smiles)
if __name__ == "__main__":
# 连接Tair数据库,并创建分子结构的向量索引,命名为“MOLSEARCH_TEST”。
tair = get_tair()
INDEX_NAME = "MOLSEARCH_TEST"
create_index()
# 写入示例数据。
do_load("D:\Test\Compound_168000001_168500000.smi")
# 在MOLSEARCH_TEST索引中,查询与"CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1"最为相似的10个分子结构。
do_search("CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1",10)本示例的正确执行结果如下:
create index done
load num 10000
load done
与查询目标分子结构最为相似的10个分子结构如下:
b'168000009' 0.0 ['CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1']
b'168003114' 29534.0 ['Cc1cc(C)cc(N2CCN(CC(=O)NC3CCCC3)C(=O)C2=O)c1']
b'168000210' 60222.0 ['COc1ccc(N2CCN(CC(=O)Nc3cc(C)cc(C)c3)C(=O)C2=O)cc1OC']
b'168001000' 61123.0 ['COc1ccc(N2CCN(CC(=O)Nc3ccc(C)cc3)C(=O)C2=O)cc1OC']
b'168003038' 64524.0 ['CCN1CCN(c2cc(C)cc(C)c2)C(=O)C1=O']
b'168003095' 67591.0 ['O=C(CN1CCN(c2cccc(Cl)c2)C(=O)C1=O)NC1CCCC1']
b'168000396' 70376.0 ['COc1ccc(N2CCN(Cc3ccc(C)cc3)C(=O)C2=O)cc1OC']
b'168002227' 71121.0 ['CCOC(=O)CN1CCN(C2CC2)C(=O)C1=O']
b'168000441' 73197.0 ['Cc1cc(C)cc(NC(=O)CN2CCN(c3ccc(F)c(F)c3)C(=O)C2=O)c1']
b'168000561' 73269.0 ['Cc1cc(C)cc(N2CCN(CC(=O)Nc3ccc(C)cc3C)C(=O)C2=O)c1']结果展示
您也可以将相似分子结构绘制成图片,示例如下。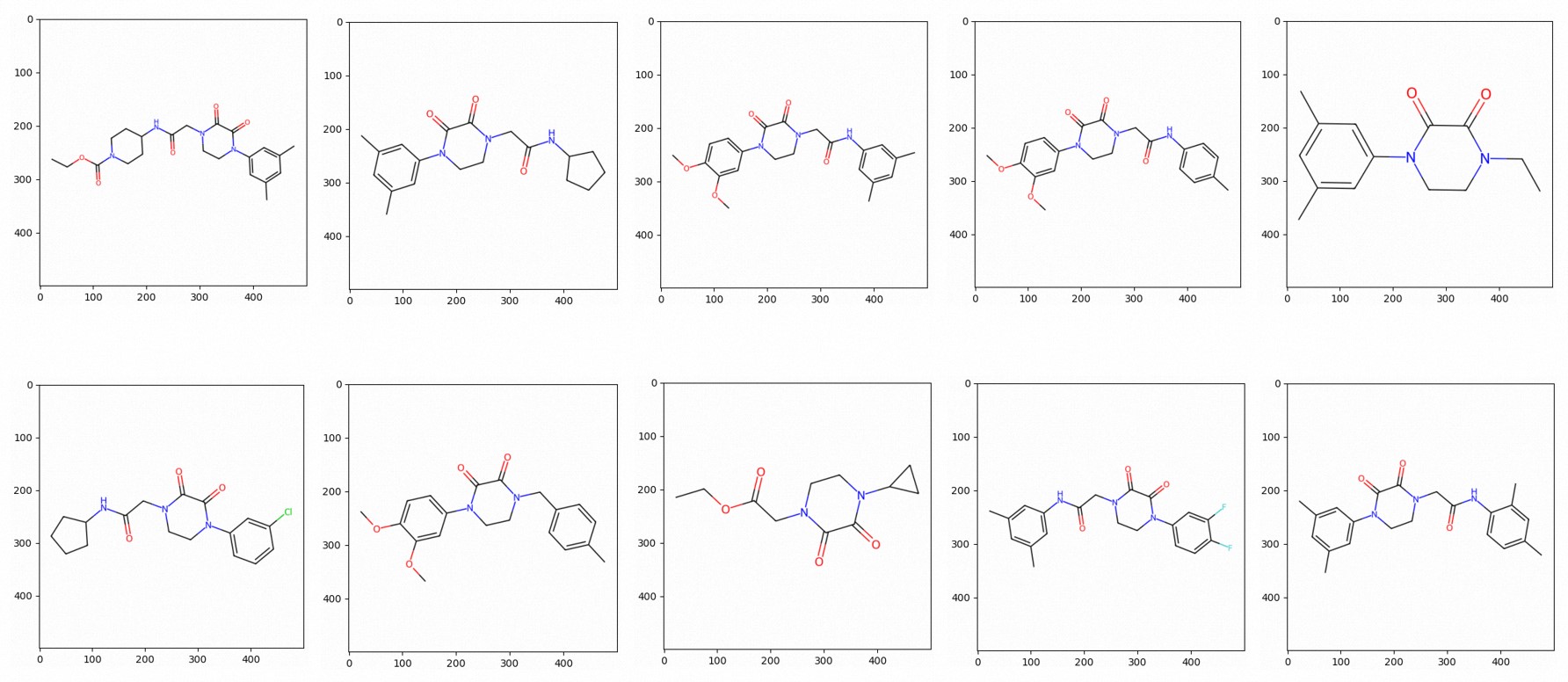
总结
使用Tair Vector检索待分析的分子结构,可以在毫秒级别检索到最相似的分子结构列表。且随着时间累积,Tair数据库中会存储越来越多的分子结构数据集,这使得后续的查询请求更加准确与及时。该方案将降低药物研发领域的研发耗时,提升整体研发效率。
